 Laboratorio de Investigación en Biología del ARN Bacteriano (LIBAB)
Laboratorio de Investigación en Biología del ARN Bacteriano (LIBAB)
Directora:
Integrantes:
Mgr. Ayala Nuñez, Teolicancihuatl
Lic. Pirajan Quintero, Ingrid Daniela
Lic. Sanhueza Salas, Luis Francisco
MD. Maia Silva, Bárbara
Est. Tevez, Maximiliano
Nuestro grupo se dedica a estudiar los ARNs no codificantes (ARNnc) y su interacción con los elementos genéticos móviles en bacterias gram negativas. Estos ARNs contribuyen en la adquisición de material genético como en la reorganización y evolución de genomas.
En nuestro laboratorio usamos como modelo de estudio a Shewanella spp., una bacteria versátil capaz de crecen en distintos hábitat, como agua, sedimentos o suelo, en condiciones extremas (bajas temperaturas, alta salinidad). Algunas especies son patógenos oportunistas del hombre, mientras que otras son comensales de invertebrados.
Entre los ARNs no codificantes que estudiamos están los sistemas CRISPR-Cas, los pequeños ARNs no codificantes (sRNA) y los intrones del grupo II:
i) Los sistemas CRISPR-Cas son considerados el sistema inmune adaptativo de las bacterias que actúan como mecanismo de defensa frente a la invasión de elementos genéticos móviles (EGM), tales como fagos y plásmidos. Nuestro grupo está enfocado a estudiar la función y estabilidad de los sistemas tipo I-F, I-E y III-B frente a diversos factores externos y celulares. En paralelo, estamos interesados en aprovechar el potencial que poseen estos sistemas como herramienta en el silenciamiento de genes mediante CRISPRi.
ii) Por otro lado, nuestro interés abarca comprender cómo ocurre la diseminación de los intrones del grupo II en la naturaleza , se conoce muy poco sobre su ocurrencia en diversos nichos. Asimismo, nos interesa evaluar la asociación de estas ribozimas con otros mecanismos de defensa bacterias, como CRISPR-Cas.
iii) Por último, estamos actualmente estudiando sRNA reguladores asociadas a virulencia. El pequeño ARN McaS es un riboregulador presente en distintos virotipos de E. coli y en Salmonella enterica, que puede ser introducido en trans de forma tal de modular la expresión de dicho gen.
Los distintos ARNs no codificantes pueden ser diseminados mediante eventos de transferencia horizontal por medio de distintos elementos genéticos móviles, como ser los elementos integrativos y conjugativos (ICE). Estas plataformas de gran tamaño poseen una gran variedad de genes con funciones diversas. Nuestro grupo se dedica a estudiar el impacto que ejerce la adquisición de grandes plataformas genéticas mediante la caracterización de la expresión y actividad de posibles ARNnc codificados en la misma y la evaluación de la respuesta del hospedador a un nuevo material invasor.
Parte de nuestro trabajo se realiza en colaboración con investigadores del Instituto Antártico Argentino para aislar y estudiar tanto intrones del grupo II como también sistemas CRISPR-Cas en bacterias de ambientes extremos, como Shewanella spp. y estudiar sus plataformas genéticas .
Colaboradores externos:
![]() Dra. Susana Vázquez - Nanobiotec, FFyB, UBA
Dra. Susana Vázquez - Nanobiotec, FFyB, UBA
![]() Dr. Walter MacCormack- Instituto Antártico Argentino
Dr. Walter MacCormack- Instituto Antártico Argentino
![]() Dr. Andrés Iriarte- Universidad de la República- Uruguay
Dr. Andrés Iriarte- Universidad de la República- Uruguay
![]() Dra. María Soledad Ramírez - California State University, Fulerton - USA
Dra. María Soledad Ramírez - California State University, Fulerton - USA
Publicaciones destacadas:
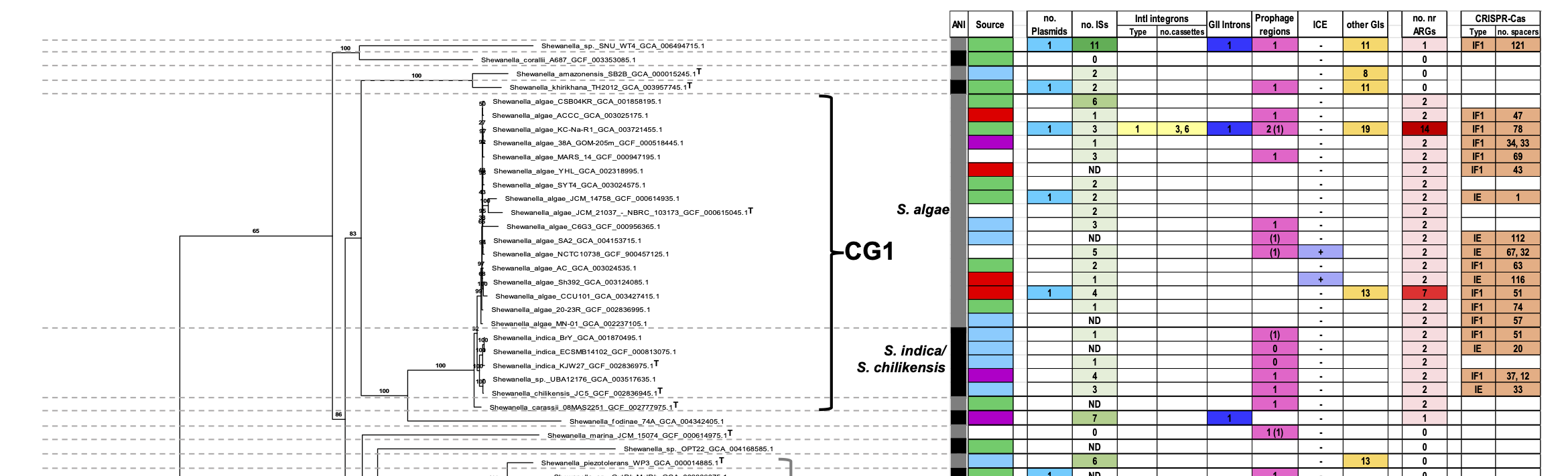
-Cerbino GN, Traglia GM, Ayala Nuñez T, Parmeciano Di Noto G, Ramírez MS, Centrón D, Iriarte A and Quiroga C (2023) Comparative genome analysis of the genus Shewanella unravels the association of key genetic traits with known and potential pathogenic lineages. Front. Microbiol. 14:1124225. doi: 10.3389/fmicb.2023.1124225
-Ayala Nuñez, T.; Cerbino, G.N.; Rapisardi, M.F.; Quiroga, C.; Centrón, D. Novel Mobile Integrons and Strain-Specific Integrase Genes within Shewanella spp. Unveil Multiple Lateral Genetic Transfer Events within The Genus. Microorganisms 2022, 10, 1102. doi:10.3390/microorganisms10061102. *co-corresponding authors.
- Parmeciano DI Noto G, Iriarte A, Ramírez MS, Centrón D, Quiroga C. ICE SXT vs. ICESh95: Co-existence of Integrative and Conjugative Elements and Competition for a New Host. Sci Rep. 2019 May 29;9(1):8045. doi:10.1038/s41598-019-44312-1
- Parmeciano Di Noto G, Molina MC, Quiroga C. Insights Into Non-coding RNAs as Novel Antimicrobial Drugs. Front Genet. 2019 Feb 22;10:57. doi: 10.3389/fgene.2019.00057.
- Wolf IR, Paschoal AR, Quiroga C, Domingues DS, de Souza RF, Pretto-Giordano LG, Vilas-Boas LA. Functional annotation and distribution overview of RNA families in 27 Streptococcus agalactiae genomes. BMC Genomics. 2018 Jul 28;19(1):556. doi: 10.1186/s12864-018-4951-z.
- Quiroga C, Nastro M, Di Conza J. Current scenario of plasmid-mediated colistin resistance in Latin America. Rev Argent Microbiol. 2019 Jan-Mar;51(1):93-100. doi: 10.1016/j.ram.2018.05.001.
- Abele D, Vazquez S, Buma AGJ, Hernandez E, Quiroga C, Held C, Frickenhaus S, Harms L, Lopez JL, Helmke E, Mac Cormack WP. Pelagic and benthic communities of the Antarctic ecosystem of Potter Cove: Genomics and ecological implications. Mar Genomics. 2017 Jun;33:1-11. doi: 10.1016/j.margen.2017.05.001.
- Parmeciano Di Noto G, Jara E, Iriarte A, Centrón D, Quiroga C. Genome analysis of a clinical isolate of Shewanella sp. uncovered an active hybrid integrative and conjugative element carrying an integron platform inserted in a novel genomic locus. Microbiology (Reading). 2016 Aug;162(8):1335-1345. doi: 10.1099/mic.0.000310.
- Parmeciano Di Noto G, Vázquez SC, MacCormack WP, Iriarte A, Quiroga C. Draft Genome of Shewanella frigidimarina Ag06-30, a Marine Bacterium Isolated from Potter Peninsula, King George Island, Antarctica. Genome Announc. 2016 May 5;4(3):e00289-16. doi: 10.1128/genomeA.00289-16.
- Quiroga C, Kronstad L, Ritlop C, Filion A, Cousineau B. Contribution of base-pairing interactions between group II intron fragments during trans-splicing in vivo. RNA. 2011 Dec;17(12):2212-21. doi: 10.1261/rna.028886.111.
- Quiroga C, Centrón D. Using genomic data to determine the diversity and distribution of target site motifs recognized by class C-attC group II introns. J Mol Evol. 2009 May;68(5):539-49. doi: 10.1007/s00239-009-9228-3.
- Quiroga C, Roy PH, Centrón D. The S.ma.I2 class C group II intron inserts at integron attC sites. Microbiology (Reading). 2008 May;154(Pt 5):1341-1353. doi: 10.1099/mic.0.2007/016360-0.
Trabajos destacados:
- Mención especial a la comunicación oral de la sección Microbiología Molecular “Interference and editing of plasmids by a type I-F1 CRISPR-Cas system in different bacteria”. Molina MC, Quiroga C. Congreso XVII SAMIGE 2022. 25 al 28 de Octubre. Los Cocos, Argentina.
- Mención especial a la comunicación oral “Análisis de elementos integrativos y conjugativos de tipo SXT/R391 presentes en genomas de bacterias marinas”. Pirajan D, Vázquez S, Quiroga C. V CAMAyA. 15 al 17 de Septiembre. La Plata, Argentina. 2021.
- Mención especial al poster “Estructura y adquisición de los módulos CRISPR-Cas en genomas de Shewanella”. Ayala Nuñez T, Cerbino G, Parmeciano Di Noto G, Marquez Perea L, Centrón D, Iriarte A, Quiroga C. V CAMAyA. 15 al 17 de Septiembre. La Plata, Argentina. 2021.
Presentaciones a Congresos
XVIII Congreso de la Sociedad Argentina de Microbiología General
Chapadmalal, Buenos Aires, Argentina, 2 al 5 de Octubre de 2023
- IDENTIFICATION AND CO-OCCURRENCE OF DEFENSE SYSTEMS IN Shewanella PANGENOME
- IN-SILICO AND EXPERIMENTAL ANALYSES OF INTEGRATIVE AND CONJUGATIVE ELEMENTS OF THE SXT/R391 TYPE 1 FAMILY REVEAL A HIGHLY VARIABLE GENE CONTENT AND A SPECIES-SPECIFIC DISSEMINATION
- UNVEILING CRISPR-Cas SYSTEMS DIVERSITY AND LOCALIZATION IN Shewanella GENOMES
- UNVEILING CRISPR-CAS I-F1 SYSTEM MOBILIZATION MECHANISM IN Shewanella xiamenensis Sh95